R doParallel: چگونه محاسبات جدول دادههای R را موازی کنیم
توزیع محاسبات را در جدول دادههای R موازی کردن راهی مطمئن برای کاهش زمان محاسباتی خطوط داده خود است. البته، این پیچیدگی بیشتری به کد اضافه میکند، اما میتواند هزینه محاسباتی شما را به طور چشمگیری کاهش دهد، به خصوص اگر همه چیز را در ابر انجام میدهید.
بسته R doParallel یک افزایش سرعت قابل توجهی به محاسبات جدول داده شما اضافه میکند و در عین حال کمترین تغییرات کد را در مقابل محاسبات جدول داده شما اعمال میکند. این بسته تمام چیزهایی که شما نیاز دارید و حتی بیشتر برای وارد شدن به دنیای موازیسازی جدول داده دارد، و امروز شما همه چیز را درباره آن خواهید آموخت. بعد از خواندن، خواهید دانست چه تغییراتی باید انجام دهید تا کد خود را به صورت موازی اجرا کنید، و چگونه تعداد هستههای پردازنده شما زمان کل محاسبه و زمان هدررفت (آمادهسازی اولیه) را تحت تأثیر قرار میدهد.
مباحث پیشنویس:
چگونگی شروع کار با R doParallel
مبنایی - چقدر R تکنخی کند است؟
عملکرد R doParallel در عمل - چگونگی موازیسازی انجامهای جدول داده
موازیسازی جدول داده R - زمان محاسبه با افزایش تعداد هستههای پردازنده کاهش مییابد؟
جمعبندی R doParallel برای جداول دادهچگونگی شروع کار با R doParallel
مطالعهی مبنایی ما درباره موازیسازی قبلاً تئوریهای اساسی و دلایلی را که باید در مورد آن مراقبت کنید پوشش داد. اگر با این مفاهیم آشنا نیستید، این مقاله فرض میکند که شما درک اساسی از مفاهیم موازیسازی R را دارید.
ما اینجا خود را تکرار نمیکنیم، اما برای خلاصه:
بسته R doParallel با استفاده از بسته foreach امکان محاسبهی موازی را فراهم میکند. این به شما امکان میدهد تا حلقههای foreach را به صورت موازی اجرا کنید، و محاسبات را بر روی چندین هستهی پردازنده انجام دهید.
برای جداول داده R، این به معنای آن است که باید آنها را به قطعات تقسیم کنید، جایی که تعداد قطعات مساوی با تعداد هستههای پردازندهای است که خوشه doParallel شما بر روی آن اجرا میشود.اگر این بستهها را نصب نکردهاید، اطمینان حاصل کنید که دستورات زیر را از کنسول R خود اجرا کنید:
install.packages(c(“foreach”, “doParallel”))
و این همه! شما آماده به کار هستید!
اجازه دهید با تنظیم یک مبنا ادامه دهیم – دیدن عملکرد R در تجمیع دادهها در یک مجموعه داده به نوعی بزرگ.
مبنایی – چقدر R تکنخی کند است؟
حالا وارد مطالب جذاب میشویم! اولین کار این است که ببینیم چگونه R در تجمیع مجموعه داده عمل میکند، که این را با استفاده از pplyr به صورت تکنخی انجام خواهیم داد.
برای این کار، یک مجموعه د
اده با ۱۰ میلیون ردیف ایجاد میکنیم. اگر اینجا همراه ما هستید، این کد را اجرا کنید:
library(dplyr)
library(stringi)
library(cleaner)
library(lubridate)
n <- 10000000
data <- data.frame(
id = 1:n,
dt = rdate(n, min = “2000-01-01”, max = “2024-01-01”),
str = stri_rand_strings(n, 4),
num1 = rnorm(n),
num2 = rnorm(n, mean = 10, sd = 2),
num3 = rnorm(n, mean = 100, sd = 10)
)
head(data)
به این کمی زمان بدهید، اما این خروجی است که باید ببینید:
Image 1 – سرآغاز مجموعه داده ۱۰ میلیون ردیفی سفارشی ما
هسته مرکزی عملکرد امروز ما مقایسهی زمانهای محاسباتی است، بنابراین ما همچنین یک تابع کمکی به نام time_diff_seconds()
را تعریف میکنیم که تفاوت زمانی بین دو زمان را در ثانیه برمیگرداند:
time_diff_seconds <- function(t1, t2) {
return(as.numeric(difftime(t1, t2, units = “secs”)))
}
حالا همه چیزی که برای پیدا کردن این که R به صورت پیشفرض چقدر کند است لازم است داریم.
R dplyr – اجرای تکنخی
بسیاری از توسعهدهندگان R از dplyr
استفاده میکنند، یک بسته که پردازش داده را آسان میکند. این سریعترین روش نیست، بنابراین ما یک راهکار دیگر را در بخش بعدی بررسی خواهیم کرد.
هدف اینجا گروهبندی مجموعه داده بر اساس ستون str
و محاسبه میانگین برای همهی ستونهای عددی است. امری آسان، البته، اما به دلیل مقدار ردیفها (۱۰ میلیون) زمان زیادی میگیرد:
dplyr_bench <- function(dataset) {
start_time <- Sys.time()
res <- dataset %>%
group_by(str) %>%
summarize(
mean_num1 = mean(num1),
mean_num2 = mean(num2),
mean_num3 = mean(num3)
)
end_time <- Sys.time()
duration <- time_diff_seconds(end_time, start_time)
print(paste0(“Total time:”, duration))
return(res)
}
dplyr_bench_res <- dplyr_bench(data)
این خروجیای است که بعد از اجرای کد فوق خواهید دید:
Image 2 – زمان کل بنچمارک dplyr
خلاصهی طولانی را کوتاه میکنیم. زمانی زیادی میگیرد. موازیسازی یک گزینه خوب به نظر میرسد، اما آیا تنها یک گزینه است؟ بیایید ببینیم اگر ما به سادگی backend
dplyr
را تعویض کنیم چه اتفاقی میافتد.
R dtplyr – اجرای dplyr با یک Backend مختلف
بست
ه R dtplyr از backend
data.table استفاده میکند که باعث میشود نتایج سریعتری را جمعآوری کند. زمان کلی محاسبهی نهایی بسیار به نوع گروهبندیای که انجام میدهید بستگی دارد، اما به طور میانگین تقریباً مطمئن هستید که زمان محاسبه کمتری خواهید داشت.
بهترین قسمت – این بسته از دستورالعملهای شبیه dplyr استفاده میکند، بنابراین تغییرات کدی که باید انجام دهید حداقل است. تنها چیز مهمی که باید به آن یادآور شوید تبدیل دادههای data.frame به tibble
است، بقیه به نسبت خودکار هستند:
library(dtplyr)
library(data.table)
dtplyr_bench <- function(dataset) {
start_time <- Sys.time()
res <- dataset %>%
lazy_dt() %>%
group_by(str) %>%
summarize(
mean_num1 = mean(num1),
mean_num2 = mean(num2),
mean_num3 = mean(num3)
) %>%
as_tibble()
end_time <- Sys.time()
duration <- time_diff_seconds(end_time, start_time)
print(paste0(“Total time:”, duration))
return(res)
}
dtplyr_dataset <- as_tibble(data)
dtplyr_bench_res <- dtplyr_bench(dtplyr_dataset)
حالا آمادهاید برای نتایج؟ دستیار خودتان را به دست بگیرید فقط بهتر است:
Image 3 – زمان کل بنچمارک dtplyr
بله، شما درست میخوانید. dtplyr برای این محاسبه ساده ۲۰ برابر سریعتر از dplyr است. این تفاوت همیشه این قدر نخواهد بود، اما این موضوع را درک میکنید – راههایی وجود دارد که R را بدون موازیسازی سریعتر کنید.
حالا ما نتایج پایه را داریم، پس بیایید ببینیم آیا R doParallel روی یک جدول داده میتواند زمان محاسبه را بیشتر کاهش دهد.
R doParallel در عمل – چگونگی موازیسازی انجامهای جدول داده
حالا به دنیای پردازش موازی R خواهیم رفت، هم با پشتیبانی از backend
dplyr
و هم dtplyr
. اگر مقاله قبلی ما درباره R doParallel را خواندهاید، میدانید که R به یک خوشه برای انجام کارهایش نیاز دارد. یک شیوهی پیشنهادی این است که هر چه بیشتر هستههایی که میتوانید به آن بدهید را به آن بدهید. ماشین ما ۱۲ هسته دارد، و ۱۱ هسته را به خوشه اختصاص میدهیم.
سپس، مجموعه داده باید به قطعاتی تقسیم شود. شما تعداد قطعاتی خواهید داشت که همان تعداد هستههایی است که به خوشه اختصاص دادهاید.
سپس، میتوانید از تابع foreach()
استفاده کنید تا تابع تجمیع داده خود را به قطعات داده، همه روی هستههای مجزا اجرا کنید.
بیایید ببینیم این با dplyr و dtplyr چگونه کار میکند.
موازیسازی جدول داده R با dplyr
تابع dplyr_parallel_bench()
مسئولیت تنظیم خوشه و اجرای تابع agg_function()
را به صورت موازی دارد. همچنین ما زمان اجرا را نگه میداریم، تا بتوانیم بررسی کنیم چه مقدار زمان توسط محاسبات گرفته شده و چه مقدار توسط آمادهسازی خوشه (ایجاد یک خوشه و تقسیم داده
) گرفته شده است:
library(doParallel)
dplyr_parallel_bench <- function(dataset, clusters) {
start_time <- Sys.time()
cl <- makeCluster(clusters)
registerDoParallel(cl)
res <- foreach(i = 1:clusters) %dopar% {
data_chunk <- dataset[((i-1)n/clusters + 1):(in/clusters), ]
agg_function(data_chunk)
}
stopCluster(cl)
res <- do.call(rbind, res)
end_time <- Sys.time()
duration <- time_diff_seconds(end_time, start_time)
print(paste0(“Total time:”, duration))
return(res)
}
حالا به یک نمونهی agg_function()
به نام dplyr_agg_function() نیاز داریم که برای هر قطعه از دادهها فراخوانده میشود. این عملکرد تقریباً همان است که ما برای dplyr_bench()
استفاده کردهایم، با این تفاوت که به جای بررسی کل داده، ما فقط به قطعهای از دادهها مینگریم:
dplyr_agg_function <- function(dataset_chunk) { res <- dataset_chunk %>%
group_by(str) %>%
summarize(
mean_num1 = mean(num1),
mean_num2 = mean(num2),
mean_num3 = mean(num3)
)
return(res)
}
حالا ما آمادهایم برای اجرای موازی دادهها در dplyr:
clusters <- 11 # Number of cores – 1
dplyr_parallel_res <- dplyr_parallel_bench(data, clusters)
این نتیجهای است که بعد از اجرای کد بالا خواهید دید:
Image 4 – زمان کل بنچمارک dplyr موازیسازیشده
عملکرد موازیسازی نه تنها برای این حالت خاص کار کرده است، بلکه همچنین به صورت چشمگیری زمان محاسبه را کاهش داده است. اما حالا آن را با dtplyr امتحان کنید.
موازیسازی جدول داده R با dtplyr
برای این کار، تابع dtplyr_parallel_bench()
را برای استفاده از backend data.table
باید ایجاد کنید. این کاملاً مشابه قبلی است، با این تفاوت که ما از توابع dtplyr
استفاده میکنیم:
dtplyr_parallel_bench <- function(dataset, clusters) {
start_time <- Sys.time()
cl <- makeCluster(clusters)
registerDoParallel(cl)
res <- foreach(i = 1:clusters) %dopar% {
data_chunk <- dataset[((i-1)n/clusters + 1):(in/clusters), ]
dtplyr_agg_function(data_chunk)
}
stopCluster(cl)
res <- do.call(rbind, res)
end_time <- Sys.time()
duration <- time_diff_seconds(end_time, start_time)
print(paste0(“Total time:”, duration))
return(res)
}
به عنوان تابع agg_function
برای استفاده در موازیسازی dtplyr، ما یک نمونه از dtplyr_agg_function()
استفاده میکنیم که تقریباً همان است که قبلاً برای dplyr_agg_function
استفاده کردیم:
dtplyr_agg_function <- function(dataset_chunk) { res <- dataset_chunk %>%
lazy_dt() %>%
group_by(str) %>%
summarize(
mean_num1 = mean(num1),
mean_num2 = mean(num2),
mean_num3 = mean(num3)
) %>%
as_tibble()
return(res)
}
حالا ما آمادهایم برای اجرای موازی دادهها در dtplyr:
clusters <- 11 # Number of cores – 1
dtplyr_parallel_res <- dtplyr_parallel_bench(data, clusters)
این نتیجهای است که بعد از اجرای کد بالا خواهید دید:
Image 5 – زمان کل بنچمارک dtplyr موازیسازیشده
به نظر میرسد که موازیسازی در dtplyr هم عملکرد بسیار خوبی دارد. از اینجا، شما میتوانید از هر دو رویکرد dplyr و dtplyr
برای موازیسازی جدول داده استفاده کنید، و از مزایایی که هر کدام ارائه میدهند بهرهبرداری کنید.
موازیسازی جدول داده R – زمان محاسبه با افزایش تعداد هستههای پردازنده کاهش مییابد؟
در حالتهای مختلف، زمان محاسبه شده ممکن است با افزایش تعداد هستههای پردازنده کاهش یابد. اما در بعضی موارد، افزایش تعداد هستهها ممکن است به یک نقطه اشباع برسد و زمان محاس
به دیگر کاهش پیدا نکند. این به زمینهی مسئله و نوع عملیاتی که انجام میدهید بستگی دارد.
برخی از موارد کاربردی که میتوانند با افزایش تعداد هستهها بهبود پیدا کنند عبارتند از:
- پردازش موازی مستقل: در این موارد، تکلیفها یکدیگر را تحت تاثیر قرار نمیدهند و انجام یک تکلیف در هر هسته به معنای کاهش زمان است. مثالهایی از این دسته شامل پردازشهای آماری مستقل مثل توابع aggregate یا summary هستند.
- مواردی که امکان جداسازی دادهها و پردازش مستقل آنها وجود دارد. در این موارد، هر قطعه از دادهها میتواند بصورت مستقل پردازش شود و در نهایت نتایج جمعآوری شوند.
در غیر این صورت، تعداد هستهها ممکن است به زودی به مرز بیشینهی سرعت موازیسازی برسد. به عنوان مثال، اگر یک عملیات به یک منبع مشترک دسترسی داشته باشد و درخواستها به یکدیگر وابسته باشند، موازیسازی به همراه افزایش تعداد هستهها ممکن است بهبودی در کارایی نداشته باشد.
بنابراین، پیش از اینکه به افزایش تعداد هستهها فکر کنید، نیاز است موارد کاربردی خود را و شرایط محیطی را مورد بررسی دقیق قرار دهید تا اطمینان حاصل شود که افزایش تعداد هستهها واقعاً زمان محاسبه را کاهش میدهد.
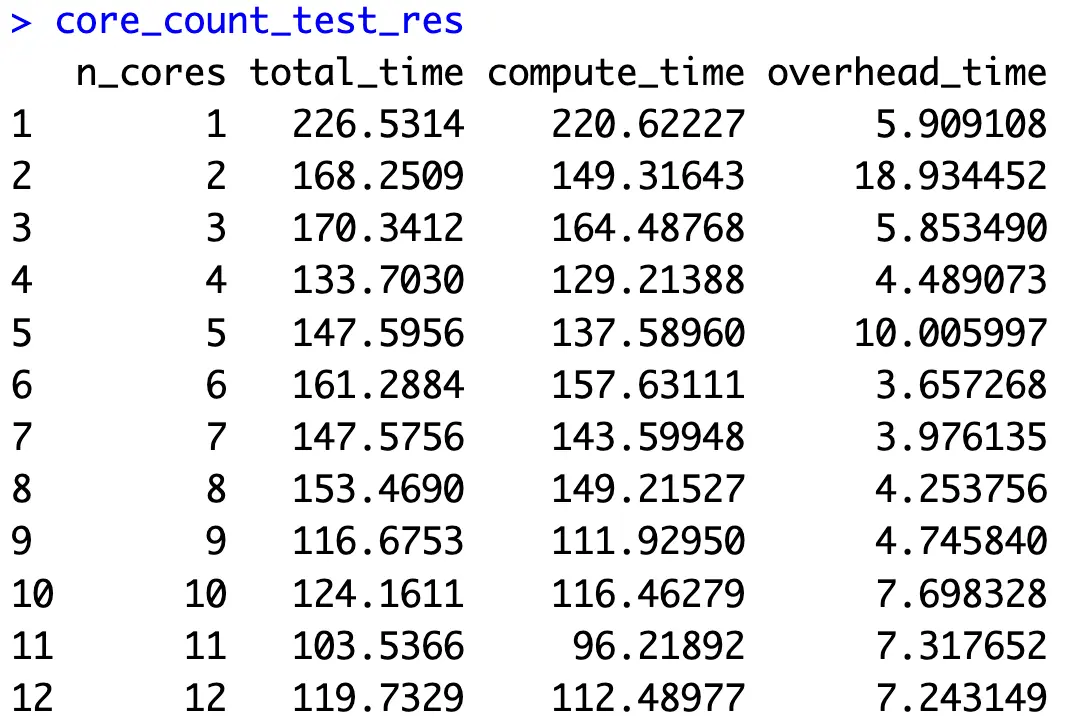
اجرای قطعه کد فوق ممکن است برای مدتی زمان برداشته، بستگی به پیکربندی سختافزاری شما دارد. در ادامه، نتایجی که دریافت کردیم عرضه میشوند:
تصویر 6 – تفاوت زمان اجرا بر اساس تعداد هستههای پردازنده
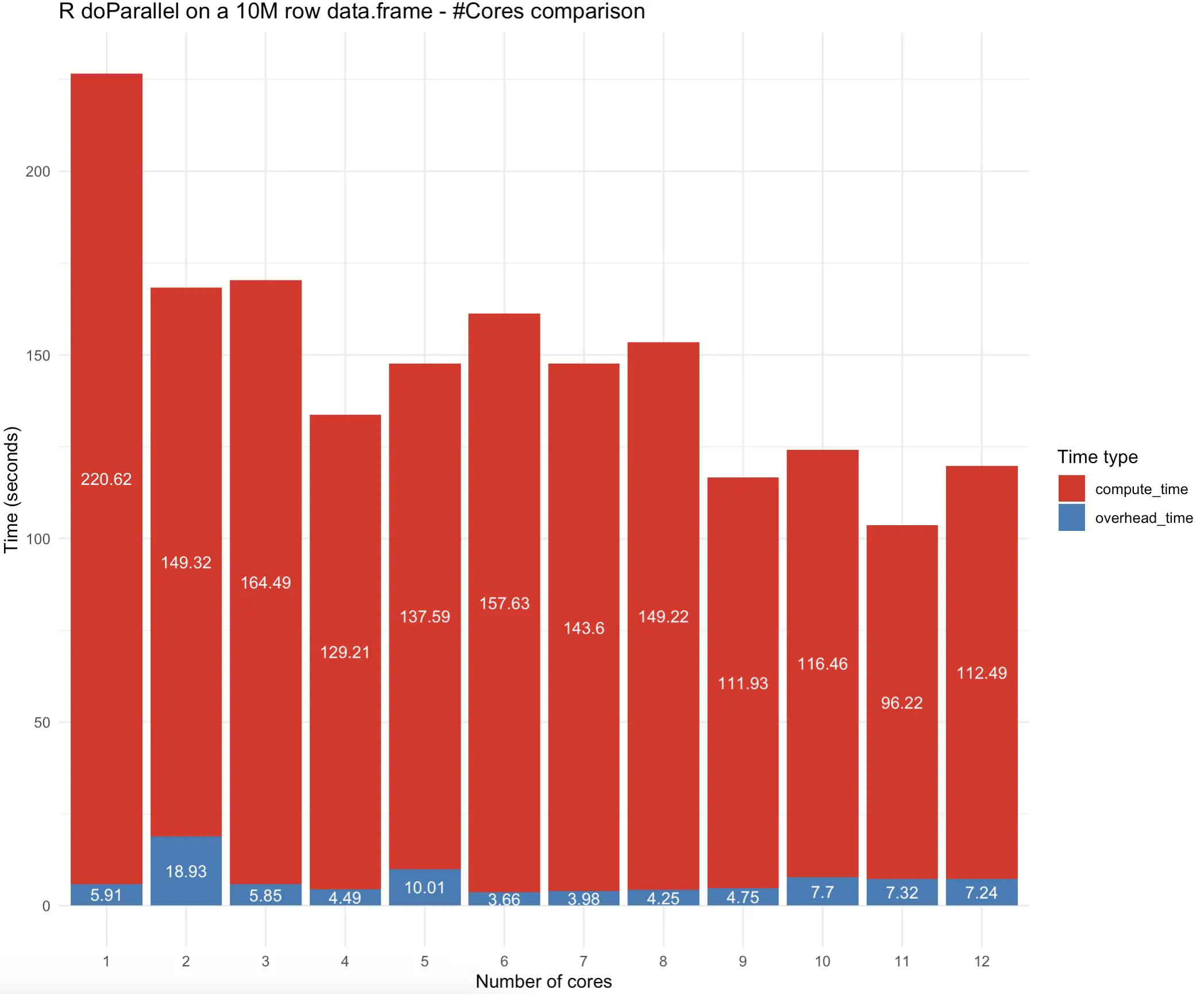
به نظر میرسد که استفاده از 11 هسته در مورد ما بهترین نتیجه را داشته است، اما بیایید نتایج را به صورت تصویری بررسی کنیم تا ببینیم آیا الگوهایی مشخص میشود: تصویر 7 – تفاوت زمان اجرا بر اساس تعداد هستههای پردازنده (نمودار)
به طور خلاصه، استفاده از 11 هسته پردازنده نتایج را سریعتر به دست آورد، اما پیادهسازی با 4 هسته نیز به طور چشمگیری عقب نماند. مهم است که توجه داشته باشید که کاهش زمان محاسباتی با افزایش تعداد هستهها به صورت خطی نیست و گاهی اوقات حتی معنیدار نیست.
آیا میخواهید بدانید چگونه این نمودار ستونی جذاب را ایجاد کردیم؟ اینجا یک راهنمایی برای تجسم داده با R و ggplot2 است.
در ادامه، یک خلاصه کوتاه ارائه میدهیم.
خلاصه کردن R doParallel برای فریمهای داده
در R، معمولاً همواره پردازش موازی پاسخی برای اجرای سریعتر کدهای شماست. با این حال، گاهی اوقات این پاسخ صحیح نیست زیرا کد پیچیدهتری برای نوشتن دارد. حتی اگر شما به این مورد اهمیت ندهید، ممکن است راهحلهای سادهتری وجود داشته باشد که نیازی به موازیسازی ندارند.
این نکته امروز به شکل کامل مشخص شد. پیادهسازی ساده dtplyr در R سریعتر از هر چیزی بود که موازیسازی ارائه کرد. اما این ممکن است برای شما صدق نکند. همیشه مهم است که تمام حالات را بر روی کد خود تست کنید، زیرا عملیات دادهای شما ممکن است در پیچیدگی متفاوت باشد.